您现在的位置是:首页 >技术交流 >Ubuntu下跑通 nnUNet v2网站首页技术交流
Ubuntu下跑通 nnUNet v2
网上关于nnUNet运行的教程大部分是针对nnUNet v1的。但由于nnUNet v2已经推出,而且相对于v1有了很大的更新。所以个人只能啃nnUNet的英文文档+参考在Windows上实现nnU-Net v2的环境配置_netv2_无聊的程序猿的博客-CSDN博客 实现了代码的复现。
1.System requirements及笔者的基础环境:
系统要求:Linux (Ubuntu 18.04, 20.04, 22.04; centOS, RHEL), Windows and MacOS
硬件要求:GPU, CPU and Apple M1/M2 (苹果设备不支持3D 卷积, 需要使用CPU).
训练硬件要求:至少有10G显存的GPU;至少6核的CPU
环境要求:python 3.9及以上
笔者的基础环境:
系统:Ubuntu 20.04 python:3.10.9 pytorch:2.0.0 cuda:11.7
2.数据准备:
2.1 下载代码:命令行运行git clone https://github.com/MIC-DKFZ/nnUNet.git
2.2 conda create nnUNet 并 activate nnUNet
2.3 进到代码所在的目录下,安装环境 cd nnUNet
pip install -e .
2.4 新建几个文件夹,新手可以参考我创建的文件夹目录结构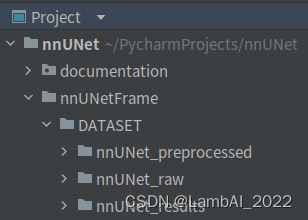
2.5 添加数据目录到系统环境变量中:运行sudo gedit /home/用户名/.bashrc,在结尾根据自己数据的目录,添加三行内容,例如我的数据文件所在目录如3.4,我添加的内容如下:
export nnUNet_raw="/home/lang/PycharmProjects/nnUNet/nnUNetFrame/DATASET/nnUNet_raw"
export nnUNet_preprocessed="/home/lang/PycharmProjects/nnUNet/nnUNetFrame/DATASET/nnUNet_preprocessed"
export nnUNet_results="/home/lang/PycharmProjects/nnUNet/nnUNetFrame/DATASET/nnUNet_results"
2.6 到Medical Segmentation Decathlon中下载数据集,可以在任何一个路径path下,不要放到步骤2.5的目录中。
2.7 数据转化,在命令行输如下命令,把3.6路径下的数据集进行转化
nnUNetv2_convert_MSD_dataset -i 步骤2.6数据集的路径 -overwrite_id 201
说明:201是数据集的ID,如果是自定义数据集,10以上都可以。
转换后,会看到nnUnet_raw目录下产生了新的文件。

2.8 数据集传入到nnUNet_preprocessed目录下:
nnUNetv2_plan_and_preprocess -d DATASET_ID --verify_dataset_integrity
参数说明:DATASET_ID是数据的ID,首次运行建议加--verify_dataset_integrity参数,对数据进行正确性检查。
我运行的命令是:nnUNetv2_plan_and_preprocess -d 201 --verify_dataset_integrity
注意:这里-d参数值需要与3.7的ID一致,
运行后nnUNet_preprocessed文件夹下产生了新的文件

3.开始训练
到/nnUNet/nnunetv2/training/nnUNetTrainer/nnUNetTrainer.py中,可修改self.num_epochs,减少训练epoch
nnUNetv2_train DATASET_NAME_OR_ID UNET_CONFIGURATION FOLD [additional options, see -h]
参数说明:DATASET_NAME_OR_ID是数据集ID UNET_CONFIGURATION是数据格式 FOLD代表选择五折交叉验证中的哪个文件夹做训练,取值范围[0,1,2,3,4],UNET_CONFIGURATION参数是数据类型参数,默认是2d, 其他可选值3d_fullres, 3d_lowres, 3d_cascade_lowres。
我运行的命令是:nnUNetv2_train 201 3d_fullres 4
几个坑:
1.如果pycharm是从软件商店安装的,它在运行时使用的环境变量文件不是系统环境变量/home/username/bashrc;因此应该直接从pycharm官网下载pycharm安装包,解压后通过命令行运行bin目录下的./pycharm.sh文件启动。
2.如果仅仅通过conda创建了一个空虚拟环境,在pycharm里是无法切换到这个空环境的。必须将必要的package install到虚拟环境中才可以。
3.如果使用conda时报异常,排除其他原因无果后,可以查看是否开启了VPN。将VPN关闭,conda功能恢复正常。





 U8W/U8W-Mini使用与常见问题解决
U8W/U8W-Mini使用与常见问题解决 QT多线程的5种用法,通过使用线程解决UI主界面的耗时操作代码,防止界面卡死。...
QT多线程的5种用法,通过使用线程解决UI主界面的耗时操作代码,防止界面卡死。... stm32使用HAL库配置串口中断收发数据(保姆级教程)
stm32使用HAL库配置串口中断收发数据(保姆级教程) 分享几个国内免费的ChatGPT镜像网址(亲测有效)
分享几个国内免费的ChatGPT镜像网址(亲测有效) Allegro16.6差分等长设置及走线总结
Allegro16.6差分等长设置及走线总结